


/中文/

/中文/

/中文/

/中文/

/中文/

/中文/

/中文/

/中文/

/中文/

/中文/
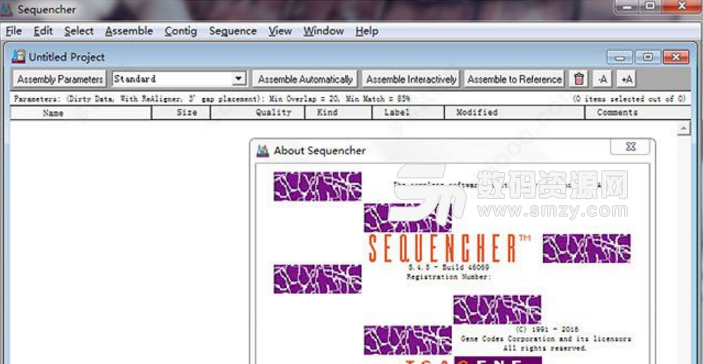
要是需要对dna来进行分析相关的软件,用Sequencher最新版就很合适哦,支持来进行序列的装配、序列编辑以及对数据来进行自动分析等多种强大的功能哦,这款DNA序列拼接分析软件还能进行矢量调整、调整矢量等功能,有了Sequencher最新版会让你工作起来更方便哦!
自动分析
Sequencher 可对你的数据进行批处理,这个过程是透明、用户可定义、可恢复的,并且 Sequencher 从不为了自动化的目的而破坏你科学结论的正确性。
当你工作在多个序列时,Sequencher 可以:
调用第二峰
调整矢量
调整低质量的尾端
创建一致序列
恢复到实验数据
Sequencher 总是管理两份数据,一份编辑过的,一份则是原始的导入数据。当你应用“Revert to Experimental Data”命令到在你项目中已选定的序列或者在一序列中的选中部分时,你可以撤销所有或者部分编辑动作。
自从 4.5 以后,Sequencher 就包含了一个新的自动化工具,“Assemble by Name”。依靠“Assemble by Name”,你可以选择片段名的一部分作为一个共享的标识符,或者说是“装配句柄”。Sequencher 就可以将选择和名字自动转换为 Contig。例如,仅仅通过一个按钮的点击,你就可以将 90 个文件,45对前端和反向序列转换成 45 个根据你的病人编号命名的 Contig。装配参数的每个变化都会重组你的片段,因此你可以根据克隆编号、日期、引物,或者其他任何你记录在序列名称中的特征,来装配 Contig。
矢量调整
自动排序器以基础调用错误生成结果。从 DNA 库克隆的序列通常包括矢量序列、polyA 尾,或其他不相关序列。内含子和引物序列通常会和放大的外显子序列侧面相连。不调整的话,这些污染物会扭曲你的装配和下游分析。 Sequencher 允许你调整低质量或者含糊的数据。“Trim Ends”将令人误解的数据从序列片段的尾部去除。“Trim Vector”将污染序列尾部的特定序列数据移除。“Trim to Reference”剔除超出装配参考序列的序列尾部。
SNP 检测
使用 Sequencher 来进行比较对准以便鉴别和报告 SNP 以及突变。例如,调用第二峰..功能分析你的所有序列以寻找潜在的杂合子。你可以控制定义一个杂合子的严格度。你可以从一个杂合子跳到下一个,而仅仅需要在 Base View 里面按一下空格键。你还可以观察一致及其参考序列的转换。一致及其参考序列的区别会列在一个可导出的表格内。参考序列确保从一个装配到下一个装配时,SNP 的数目保持一致。
序列装配
Sequencher 的装配算法可以将你的 DNA 片断快速而又准确的装配起来。自觉的工具允许你在数秒内就可以设置好参数并且调整它们。你完全可以不顾方向的装配序列。Sequencher 会比较前端和反转补体方向来装配最可能的 Contig。
序列编辑
Sequencher 能够给你足够的信心认为一个序列是绝对正确的。你可以只观察一个序列的色谱图,你也可以从前端或反方向同时查看多个对齐的色谱图。 在你的已对齐数据中滚动是很容易的。你可以使用 Sequencher 的选择工具来高亮不一致或者低质量的区域。

 小学口算生成器(口算题自动生成小程序) 最新版理科工具
/ 711K
小学口算生成器(口算题自动生成小程序) 最新版理科工具
/ 711K
 mathtype7.0中文版(附序列号) 永久授权版理科工具
/ 9M
mathtype7.0中文版(附序列号) 永久授权版理科工具
/ 9M
 mathtype 6.9b产品密钥(激活码) 免费版理科工具
/ 9M
mathtype 6.9b产品密钥(激活码) 免费版理科工具
/ 9M
 化学元素周期表高清图合集(五个版本) 最新版理科工具
/ 916K
化学元素周期表高清图合集(五个版本) 最新版理科工具
/ 916K
 胡迪数学题口算软件(制作任意四则运算口表) v2.0 绿色版理科工具
/ 14K
胡迪数学题口算软件(制作任意四则运算口表) v2.0 绿色版理科工具
/ 14K
 mathtype7.4中文注册版(数学公式编辑器)理科工具
/ 38M
mathtype7.4中文注册版(数学公式编辑器)理科工具
/ 38M
 mathtype 6.9b无限试用版(含激活码) 免费版理科工具
/ 9M
mathtype 6.9b无限试用版(含激活码) 免费版理科工具
/ 9M
 MathCAD15简体中文版理科工具
/ 291M
MathCAD15简体中文版理科工具
/ 291M
 物理tracker中文版(物理学习软件) v4.9.1 最新版理科工具
/ 35M
物理tracker中文版(物理学习软件) v4.9.1 最新版理科工具
/ 35M
 MathType6.9d中文版(含注册码激活密钥) 免费版理科工具
/ 7M
MathType6.9d中文版(含注册码激活密钥) 免费版理科工具
/ 7M
 小学口算生成器(口算题自动生成小程序) 最新版理科工具
/ 711K
小学口算生成器(口算题自动生成小程序) 最新版理科工具
/ 711K
 mathtype7.0中文版(附序列号) 永久授权版理科工具
/ 9M
mathtype7.0中文版(附序列号) 永久授权版理科工具
/ 9M
 mathtype 6.9b产品密钥(激活码) 免费版理科工具
/ 9M
mathtype 6.9b产品密钥(激活码) 免费版理科工具
/ 9M
 化学元素周期表高清图合集(五个版本) 最新版理科工具
/ 916K
化学元素周期表高清图合集(五个版本) 最新版理科工具
/ 916K
 胡迪数学题口算软件(制作任意四则运算口表) v2.0 绿色版理科工具
/ 14K
胡迪数学题口算软件(制作任意四则运算口表) v2.0 绿色版理科工具
/ 14K
 mathtype7.4中文注册版(数学公式编辑器)理科工具
/ 38M
mathtype7.4中文注册版(数学公式编辑器)理科工具
/ 38M
 mathtype 6.9b无限试用版(含激活码) 免费版理科工具
/ 9M
mathtype 6.9b无限试用版(含激活码) 免费版理科工具
/ 9M
 MathCAD15简体中文版理科工具
/ 291M
MathCAD15简体中文版理科工具
/ 291M
 物理tracker中文版(物理学习软件) v4.9.1 最新版理科工具
/ 35M
物理tracker中文版(物理学习软件) v4.9.1 最新版理科工具
/ 35M
 MathType6.9d中文版(含注册码激活密钥) 免费版理科工具
/ 7M
MathType6.9d中文版(含注册码激活密钥) 免费版理科工具
/ 7M